LUYOR-3109高強(qiáng)度紫外催化光源促銷
LUYOR-3109紫外光源采用了9顆365nm大功率led,安裝有二次光學(xué)透鏡,輸出紫外線強(qiáng)度高,...
2024-08-08
LUYOR-3109紫外光源采用了9顆365nm大功率led,安裝有二次光學(xué)透鏡,輸出紫外線強(qiáng)度高,...
2024-08-08
KillerRed是個(gè)完全由基因編碼的光毒性紅色熒光蛋白,可接受綠色光照(540~580nm)生成活...
2024-04-29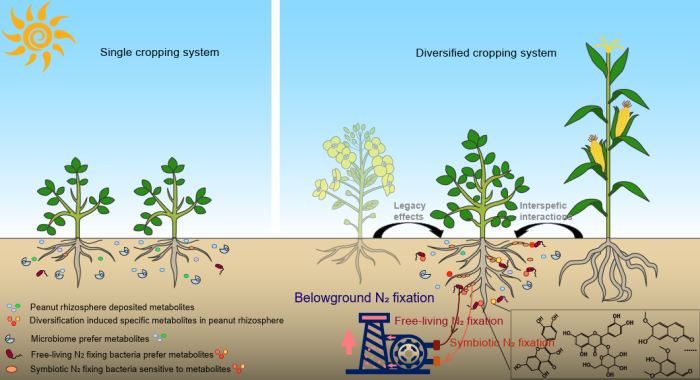
中國(guó)科學(xué)院南京土壤研究所彭新華研究員團(tuán)隊(duì)陳晏副研究員在農(nóng)田長(zhǎng)期多樣化種植下,種間植物根際對(duì)話調(diào)控土壤...
2024-04-07咨詢熱線
021-64195798作者:生命科學(xué)事業(yè)部時(shí)間:2019-09-26 08:30:24瀏覽3288 次
全基因組重測(cè)序是對(duì)基因組序列已知的個(gè)體進(jìn)行基因組測(cè)序,并在個(gè)體或群體水平上進(jìn)行差異性分析的方法。隨著基因組測(cè)序成本的不斷降低,人類疾病的致病突變研究由外顯子區(qū)域擴(kuò)大到全基因組范圍。通過(guò)構(gòu)建不同長(zhǎng)度的插入片段文庫(kù)和短序列、雙末端測(cè)序相結(jié)合的策略進(jìn)行高通量測(cè)序,實(shí)現(xiàn)在全基因組水平上檢測(cè)疾病關(guān)聯(lián)的常見(jiàn)、低頻、甚至是罕見(jiàn)的突變位點(diǎn),以及結(jié)構(gòu)變異等,具有重大的科研和產(chǎn)業(yè)價(jià)值。
1、什么是高通量測(cè)序?
高通量測(cè)序技術(shù)(High-throughputsequencing,HTS)是對(duì)傳統(tǒng)Sanger測(cè)序(稱為一代測(cè)序技術(shù))革命性的改變,一次對(duì)幾十萬(wàn)到幾百萬(wàn)條核酸分子進(jìn)行序列測(cè)定,因此在有些文獻(xiàn)中稱其為下一代測(cè)序技術(shù)(next generation sequencing,NGS )足見(jiàn)其劃時(shí)代的改變,同時(shí)高通量測(cè)序使得對(duì)一個(gè)物種的轉(zhuǎn)錄組和基因組進(jìn)行細(xì)致全貌的分析成為可能,所以又被稱為深度測(cè)序(Deep sequencing)。
2、什么是基因組重測(cè)序(Genome Re-sequencing)
全基因組重測(cè)序是對(duì)基因組序列已知的個(gè)體進(jìn)行基因組測(cè)序,并在個(gè)體或群體水平上進(jìn)行差異性分析的方法。隨著基因組測(cè)序成本的不斷降低,人類疾病的致病突變研究由外顯子區(qū)域擴(kuò)大到全基因組范圍。通過(guò)構(gòu)建不同長(zhǎng)度的插入片段文庫(kù)和短序列、雙末端測(cè)序相結(jié)合的策略進(jìn)行高通量測(cè)序,實(shí)現(xiàn)在全基因組水平上檢測(cè)疾病關(guān)聯(lián)的常見(jiàn)、低頻、甚至是罕見(jiàn)的突變位點(diǎn),以及結(jié)構(gòu)變異等,具有重大的科研和產(chǎn)業(yè)價(jià)值。
3、什么是de novo測(cè)序
de novo測(cè)序也稱為從頭測(cè)序:其不需要任何現(xiàn)有的序列資料就可以對(duì)某個(gè)物種進(jìn)行測(cè)序,利用生物信息學(xué)分析手段對(duì)序列進(jìn)行拼接,組裝,從而獲得該物種的基因組圖譜。獲得一個(gè)物種的全基因組序列是加快對(duì)此物種了解的重要捷徑。隨著新一代測(cè)序技術(shù)的飛速發(fā)展,基因組測(cè)序所需的成本和時(shí)間較傳統(tǒng)技術(shù)都大大降低,大規(guī)模基因組測(cè)序漸入佳境,基因組學(xué)研究也迎來(lái)新的發(fā)展契機(jī)和革命性突破。利用新一代高通量、率測(cè)序技術(shù)以及強(qiáng)大的生物信息分析能力,可以、低成本地測(cè)定并分析所有生物的基因組序列。
4、什么是外顯子測(cè)序(whole exon sequencing)
外顯子組測(cè)序是指利用序列捕獲技術(shù)將全基因組外顯子區(qū)域DNA捕捉并富集后進(jìn)行高通量測(cè)序的基因組分析方法。外顯子測(cè)序相對(duì)于基因組重測(cè)序成本較低,對(duì)研究已知基因的SNP、Indel等具有較大的優(yōu)勢(shì),但無(wú)法研究基因組結(jié)構(gòu)變異如染色體斷裂重組等。
5、什么是mRNA測(cè)序 (RNA-seq)
轉(zhuǎn)錄組學(xué)(tranomics)是在基因組學(xué)后新興的一門學(xué)科,即研究特定細(xì)胞在某一功能狀態(tài)下所能轉(zhuǎn)錄出來(lái)的所有RNA(包括mRNA和非編碼RNA)的類型與拷貝數(shù)。Illumina提供的mRNA測(cè)序技術(shù)可在整個(gè)mRNA領(lǐng)域進(jìn)行各種相關(guān)研究和新的發(fā)現(xiàn)。mRNA測(cè)序不對(duì)引物或探針進(jìn)行設(shè)計(jì),可自由提供關(guān)于轉(zhuǎn)錄的客觀和權(quán)威信息。研究人員僅需要一次試驗(yàn)即可快速生成完整的poly-A尾的RNA完整序列信息,并分析基因表達(dá)、cSNP、全新的轉(zhuǎn)錄、全新異構(gòu)體、剪接位點(diǎn)、等位基因特異性表達(dá)和罕見(jiàn)轉(zhuǎn)錄等最全面的轉(zhuǎn)錄組信息。簡(jiǎn)單的樣品制備和數(shù)據(jù)分析軟件支持在所有物種中的mRNA測(cè)序研究。
6、什么是small RNA測(cè)序
SmallRNA(micro RNAs、siRNAs和 pi RNAs)是生命活動(dòng)重要的調(diào)控因子,在基因表達(dá)調(diào)控、生物個(gè)體發(fā)育、代謝及疾病的發(fā)生等生理過(guò)程中起著重要的作用。Illumina能夠?qū)?xì)胞或者組織中的全部Small RNA進(jìn)行深度測(cè)序及定量分析等研究。實(shí)驗(yàn)時(shí)首先將18-30 nt范圍的Small RNA從總RNA中分離出來(lái),兩端分別加上特定接頭后體外反轉(zhuǎn)錄做成cDNA再做進(jìn)一步處理后,利用測(cè)序儀對(duì)DNA片段進(jìn)行單向末端直接測(cè)序。通過(guò)Illumina對(duì)Small RNA大規(guī)模測(cè)序分析,可以從中獲得物種全基因組水平的miRNA圖譜,實(shí)現(xiàn)包括新miRNA分子的挖掘,其作用靶基因的預(yù)測(cè)和鑒定、樣品間差異表達(dá)分析、miRNAs聚類和表達(dá)譜分析等科學(xué)應(yīng)用。
7、什么是miRNA測(cè)序
成熟的microRNA(miRNA)是17~24nt的單鏈非編碼RNA分子,通過(guò)與mRNA相互作用影響目標(biāo)mRNA的穩(wěn)定性及翻譯,最終誘導(dǎo)基因沉默,調(diào)控著基因表達(dá)、細(xì)胞生長(zhǎng)、發(fā)育等生物學(xué)過(guò)程。基于第二代測(cè)序技術(shù)的microRNA測(cè)序,可以一次性獲得數(shù)百萬(wàn)條microRNA序列,能夠快速鑒定出不同組織、不同發(fā)育階段、不同疾病狀態(tài)下已知和未知的microRNA及其表達(dá)差異,為研究microRNA對(duì)細(xì)胞進(jìn)程的作用及其生物學(xué)影響提供了有力工具。
8、什么是Chip-seq
染色質(zhì)免疫共沉淀技術(shù)(ChromatinImmunoprecipitation,ChIP)也稱結(jié)合位點(diǎn)分析法,是研究體內(nèi)蛋白質(zhì)與DNA相互作用的有力工具,通常用于轉(zhuǎn)錄因子結(jié)合位點(diǎn)或組蛋白特異性修飾位點(diǎn)的研究。將ChIP與第二代測(cè)序技術(shù)相結(jié)合的ChIP-Seq技術(shù),能夠地在全基因組范圍內(nèi)檢測(cè)與組蛋白、轉(zhuǎn)錄因子等互作的DNA區(qū)段。
ChIP-Seq的原理是:首先通過(guò)染色質(zhì)免疫共沉淀技術(shù)(ChIP)特異性地富集目的蛋白結(jié)合的DNA片段,并對(duì)其進(jìn)行純化與文庫(kù)構(gòu)建;然后對(duì)富集得到的DNA片段進(jìn)行高通量測(cè)序。研究人員通過(guò)將獲得的數(shù)百萬(wàn)條序列標(biāo)簽定位到基因組上,從而獲得全基因組范圍內(nèi)與組蛋白、轉(zhuǎn)錄因子等互作的DNA區(qū)段信息。
9、什么是CHIRP-Seq
CHIRP- Seq ( Chromatin Isolationby RNA Purification ) 是一種檢測(cè)與RNA綁定的DNA和蛋白的高通量測(cè)序方法。方法是通過(guò)設(shè)計(jì)生物素或鏈霉親和素探針,把目標(biāo)RNA拉下來(lái)以后,與其共同作用的DNA染色體片段就會(huì)附在到磁珠上,最后把染色體片段做高通量測(cè)序,這樣會(huì)得到該RNA能夠結(jié)合到在基因組的哪些區(qū)域,但由于蛋白測(cè)序技術(shù)不夠成熟,無(wú)法知道與該RNA結(jié)合的蛋白。
10、什么是RIP-seq
RNA Immunoprecipitation是研究細(xì)胞內(nèi)RNA與蛋白結(jié)合情況的技術(shù),是了解轉(zhuǎn)錄后調(diào)控網(wǎng)絡(luò)動(dòng)態(tài)過(guò)程的有力工具,能幫助我們發(fā)現(xiàn)miRNA的調(diào)節(jié)靶點(diǎn)。這種技術(shù)運(yùn)用針對(duì)目標(biāo)蛋白的抗體把相應(yīng)的RNA-蛋白復(fù)合物沉淀下來(lái),然后經(jīng)過(guò)分離純化就可以對(duì)結(jié)合在復(fù)合物上的RNA進(jìn)行測(cè)序分析。
RIP可以看成是普遍使用的染色質(zhì)免疫沉淀ChIP技術(shù)的類似應(yīng)用,但由于研究對(duì)象是RNA-蛋白復(fù)合物而不是DNA-蛋白復(fù)合物,RIP實(shí)驗(yàn)的優(yōu)化條件與ChIP實(shí)驗(yàn)不太相同(如復(fù)合物不需要固定,RIP反應(yīng)體系中的試劑和抗體不能含有RNA酶,抗體需經(jīng)RIP實(shí)驗(yàn)驗(yàn)證等等)。RIP技術(shù)下游結(jié)合microarray技術(shù)被稱為RIP-Chip,幫助我們更高通量地了解癌癥以及其它疾病整體水平的RNA變化。
11、什么是CLIP-seq
CLIP-seq,又稱為HITS - CLIP,即紫外交聯(lián)免疫沉淀結(jié)合高通量測(cè)序 ( crosslinking-immunprecipitationand high-throughput sequencing ) , 是一項(xiàng)在全基因組水平揭示RNA分子與RNA結(jié)合蛋白相互作用的革命性技術(shù)。其主要原理是基于RNA分子與RNA結(jié)合蛋白在紫外照射下發(fā)生耦聯(lián),以RNA結(jié)合蛋白的特異性抗體將RNA-蛋白質(zhì)復(fù)合體沉淀之后,回收其中的RNA片段,經(jīng)添加接頭、RT-PCR等步驟,對(duì)這些分子進(jìn)行高通量測(cè)序,再經(jīng)生物信息學(xué)的分析和處理、總結(jié),挖掘出其特定規(guī)律,從而深入揭示RNA結(jié)合蛋白與RNA分子的調(diào)控作用及其對(duì)生命的意義。
12、什么是染色體構(gòu)象捕獲技術(shù):
3C 通常是用啟動(dòng)子或者某一個(gè)基因或者基因組某一個(gè)短的片段在鄰近的幾十kb或者幾百kb基因組掃描可以獲得相互作用區(qū)域。由于實(shí)驗(yàn)需要特異性引物,因而實(shí)驗(yàn)室相當(dāng)費(fèi)力的,且檢測(cè)范圍小。
4C同3C一樣做單位點(diǎn)的檢測(cè),但其檢測(cè)擴(kuò)展到了整個(gè)基因組上。主要是引入了反向PCR,因而只需要對(duì)這一單一位點(diǎn)設(shè)計(jì)引物即可。
5C 做兩個(gè)大片段之間相互作用點(diǎn)的檢測(cè),可以達(dá)到10Mb水平。其仍需使用引物,且引物設(shè)計(jì)是其技術(shù)的難點(diǎn)。
Hi-C 可以實(shí)現(xiàn)基因組對(duì)基因組水平的檢測(cè),但是獲得高精度需要非常大的測(cè)序深度
ChIA-PET標(biāo)在于特定的蛋白因子及其相關(guān)聯(lián)的染色質(zhì)相互作用。該技術(shù)將配對(duì)末端標(biāo)簽測(cè)序技術(shù)與ChIP相結(jié)合,對(duì)富集了某種蛋白質(zhì)的DNA 片段進(jìn)行交聯(lián),可以測(cè)定全基因組范圍的特定轉(zhuǎn)錄因子參與的染色質(zhì)遠(yuǎn)程交互作用,從而可以呈現(xiàn)高特異性和高分辨率的染色質(zhì)相互作用。
13、什么是Hi-C輔助基因組組裝:
Hi-C輔助基因組組裝是指在已有二代或三代或光學(xué)圖譜輔助組裝的Draft genome序列和已知染色體數(shù)目的前提下,利用Hi-C測(cè)序數(shù)據(jù)將Draft genome序列進(jìn)行染色體群組的劃分,并確定各序列在染色體上的順序和方向,使基因組組裝組裝水平提升到染色體水平的技術(shù)。
14、什么是metagenomic(宏基因組):
Magenomics研究的對(duì)象是整個(gè)微生物群落。相對(duì)于傳統(tǒng)單個(gè)細(xì)菌研究來(lái)說(shuō),它具有眾多優(yōu)勢(shì),其中很重要的兩點(diǎn):(1) 微生物通常是以群落方式共生于某一小生境中,它們的很多特性是基于整個(gè)群落環(huán)境及個(gè)體間的相互影響的,因此做Metagenomics研究比做單個(gè)個(gè)體的研究更能發(fā)現(xiàn)其特性;(2)Metagenomics研究無(wú)需分離單個(gè)細(xì)菌,可以研究那些不能被實(shí)驗(yàn)室分離培養(yǎng)的微生物。
宏基因組是基因組學(xué)一個(gè)新興的科學(xué)研究方向。宏基因組學(xué)(又稱元基因組學(xué),環(huán)境基因組學(xué),生態(tài)基因組學(xué)等),是研究直接從環(huán)境樣本中提取的基因組遺傳物質(zhì)的學(xué)科。傳統(tǒng)的微生物研究依賴于實(shí)驗(yàn)室培養(yǎng),宏基因組的興起填補(bǔ)了無(wú)法在傳統(tǒng)實(shí)驗(yàn)室中培養(yǎng)的微生物研究的空白。過(guò)去幾年中,DNA測(cè)序技術(shù)的進(jìn)步以及測(cè)序通量和分析方法的改進(jìn)使得人們得以一窺這一未知的基因組科學(xué)領(lǐng)域。
15、什么是SNP、SNV(單核苷酸位點(diǎn)變異)
單核苷酸多態(tài)性 singlenucleotide polymorphism ,SNP或單核苷酸位點(diǎn)變異SNV。個(gè)體間基因組DNA序列同一位置單個(gè)核苷酸變異(替代、插入或缺失)所引起的多態(tài)性。不同物種、個(gè)體基因組DNA序列同一位置上的單個(gè)核苷酸存在差別的現(xiàn)象。有這種差別的基因座、DNA序列等可作為基因組作圖的標(biāo)志。人基因組上平均約每1000個(gè)核苷酸即可能出現(xiàn)1個(gè)單核苷酸多態(tài)性的變化,其中有些單核苷酸多態(tài)性可能與疾病有關(guān),但可能大多數(shù)與疾病無(wú)關(guān)。單核苷酸多態(tài)性是研究人類家族和動(dòng)植物品系遺傳變異的重要依據(jù)。在研究癌癥基因組變異時(shí),相對(duì)于正常組織,癌癥中特異的單核苷酸變異是一種體細(xì)胞突變(somatic mutation),稱做SNV。
16、什么是INDEL (基因組小片段插入)
基因組上小片段(>50bp)的插入或缺失,形同SNP/SNV。
17、什么是copy number variation(CNV):基因組拷貝數(shù)變異
基因組拷貝數(shù)變異是基因組變異的一種形式,通常使基因組中大片段的DNA形成非正常的拷貝數(shù)量。例如人類正常染色體拷貝數(shù)是2,有些染色體區(qū)域拷貝數(shù)變成1或3,這樣,該區(qū)域發(fā)生拷貝數(shù)缺失或增加,位于該區(qū)域內(nèi)的基因表達(dá)量也會(huì)受到影響。如果把一條染色體分成A-B-C-D四個(gè)區(qū)域,則A-B-C-C-D/A-C-B-C-D/A-C-C-B-C-D/A-B-D分別發(fā)生了C區(qū)域的擴(kuò)增及缺失,擴(kuò)增的位置可以是連續(xù)擴(kuò)增如A-B-C-C-D也可以是在其他位置的擴(kuò)增,如A-C-B-C-D。
18、什么是structure variation(SV):基因組結(jié)構(gòu)變異
染色體結(jié)構(gòu)變異是指在染色體上發(fā)生了大片段的變異。主要包括染色體大片段的插入和缺失(引起CNV的變化),染色體內(nèi)部的某塊區(qū)域發(fā)生翻轉(zhuǎn)顛換,兩條染色體之間發(fā)生重組(inter-chromosometrans-location)等。一般SV的展示利用Circos軟件。
19、什么是Segment duplication
一般稱為SD區(qū)域,串聯(lián)重復(fù)是由序列相近的一些DNA片段串聯(lián)組成。串聯(lián)重復(fù)在人類基因多樣性的靈長(zhǎng)類基因中發(fā)揮重要作用。在人類染色體Y和22號(hào)染色體上,有很大的SD序列。
20、什么是genotype and phenotype
既基因型與表型;一般指某些單核苷酸位點(diǎn)變異與表現(xiàn)形式間的關(guān)系。
21、什么是Read?
高通量測(cè)序平臺(tái)產(chǎn)生的短序列就稱為reads。PE125,就是讀長(zhǎng)為125bp雙端測(cè)序。
22、什么是Contig?
拼接軟件基于reads之間的overlap區(qū),拼接獲得的序列稱為Contig(重疊群),無(wú)N。
23、什么是Scaffold?
基因組de novo測(cè)序,通過(guò)reads拼接獲得Contigs后,往往還需要構(gòu)建454 Paired-end庫(kù)或Illumina Mate-pair庫(kù),以獲得一定大小片段(如3Kb、6Kb、10Kb、20Kb)兩端的序列。基于這些序列,可以確定一些Contig之間的順序關(guān)系,這些先后順序已知的Contigs組成Scaffold(含有N)。
24、什么是Contig N50?
Reads拼接后會(huì)獲得一些不同長(zhǎng)度的Contigs。將所有的Contig長(zhǎng)度相加,能獲得一個(gè)Contig總長(zhǎng)度。然后將所有的Contigs按照從長(zhǎng)到短進(jìn)行排序,如獲得Contig 1,Contig 2,Contig 3...………Contig 25。將Contig按照這個(gè)順序依次相加,當(dāng)相加的長(zhǎng)度達(dá)到Contig總長(zhǎng)度的一半時(shí),最后一個(gè)加上的Contig長(zhǎng)度即為Contig N50。舉例:Contig 1+Contig 2+ Contig 3+Contig4=Contig總長(zhǎng)度*1/2時(shí),Contig 4的長(zhǎng)度即為Contig N50。Contig N50可以作為基因組拼接的結(jié)果好壞的一個(gè)判斷標(biāo)準(zhǔn)。
25、什么是Scaffold N50?
Scaffold N50與Contig N50的定義類似。Contigs拼接組裝獲得一些不同長(zhǎng)度的Scaffolds。將所有的Scaffold長(zhǎng)度相加,能獲得一個(gè)Scaffold總長(zhǎng)度。然后將所有的Scaffolds按照從長(zhǎng)到短進(jìn)行排序,如獲得Scaffold 1,Scaffold 2,Scaffold 3...………Scaffold 25。將Scaffold按照這個(gè)順序依次相加,當(dāng)相加的長(zhǎng)度達(dá)到Scaffold總長(zhǎng)度的一半時(shí),最后一個(gè)加上的Scaffold長(zhǎng)度即為Scaffold N50。
舉例:
Scaffold 1+Scaffold 2+Scaffold 3 +Scaffold 4 +Scaffold 5=Scaffold總長(zhǎng)度*1/2時(shí),Scaffold 5的長(zhǎng)度即為Scaffold N50。Scaffold N50可以作為基因組拼接的結(jié)果好壞的一個(gè)判斷標(biāo)準(zhǔn)。
26、什么是測(cè)序深度和覆蓋度?
測(cè)序深度是指測(cè)序得到的總堿基數(shù)與待測(cè)基因組大小的比值。假設(shè)一個(gè)基因大小為2M,測(cè)序深度為10X,那么獲得的總數(shù)據(jù)量為20M。覆蓋度是指測(cè)序獲得的序列占整個(gè)基因組的比例。由于基因組中的高GC、重復(fù)序列等復(fù)雜結(jié)構(gòu)的存在,測(cè)序最終拼接組裝獲得的序列往往無(wú)法覆蓋有所的區(qū)域,這部分沒(méi)有獲得的區(qū)域就稱為Gap。例如一個(gè)細(xì)菌基因組測(cè)序,覆蓋度是98%,那么還有2%的序列區(qū)域是沒(méi)有通過(guò)測(cè)序獲得的。
27、什么是RPKM、FPKM
RPKM,ReadsPer Kilobase of exon model per Million mapped reads, is defined in thisway [Mortazavi etal., 2008]:
每1百萬(wàn)個(gè)map上的reads中map到外顯子的每1K個(gè)堿基上的reads個(gè)數(shù)。
假如有1百萬(wàn)個(gè)reads映射到了人的基因組上,那么具體到每個(gè)外顯子呢,有多少映射上了呢,而外顯子的長(zhǎng)度不一,那么每1K個(gè)堿基上又有多少reads映射上了呢,這大概就是這個(gè)RPKM的直觀解釋。
如果對(duì)應(yīng)特定基因的話,那么就是每1000000 mapped到該基因上的reads中每kb有多少是mapped到該基因上的exon的read
Total exonreads:This is the number in the column with header Total exonreads in the rowfor the gene. This is the number of reads that have beenmapped to a region inwhich an exon is annotated for the gene or across theboundaries of two exons oran intron and an exon for an annotated tran ofthe gene. For eukaryotes,exons and their internal relationships are defined byannotations of type mRNA.映射到外顯子上總的reads個(gè)數(shù)。這個(gè)是映射到某個(gè)區(qū)域上的reads個(gè)數(shù),這個(gè)區(qū)域或者是已知注釋的基因或者跨兩個(gè)外顯子的邊界或者是某個(gè)基因已經(jīng)注釋的轉(zhuǎn)錄本的內(nèi)含子、外顯子。對(duì)于真核生物來(lái)說(shuō),外顯子和它們自己內(nèi)部的關(guān)系由某類型的mRNA來(lái)注釋。
Exonlength:This is the number in the column with theheader Exon length inthe row for the gene, divided by 1000. This is calculatedas the sum of thelengths of all exons annotated for the gene. Each exon isincluded only once inthis sum, even if it is present in more annotatedtrans for the gene.Partly overlapping exons will count with their fulllength, even though theyshare the same region.外顯子的長(zhǎng)度。計(jì)算時(shí),計(jì)算所有某個(gè)基因已注釋的所有外顯子長(zhǎng)度的總和。即使某個(gè)基因以多種注釋的轉(zhuǎn)錄本呈現(xiàn),這個(gè)外顯子在求和時(shí)只被包含一次。即使部分重疊的外顯子共享相同的區(qū)域,重疊的外顯子以其總長(zhǎng)來(lái)計(jì)算。
Mapped reads: The sum of all the numbers in the column with header Totalgenereads. The Total gene reads for a gene is the total number ofreads that aftermapping have been mapped to the region of the gene. Thus thisincludes all thereads uniquely mapped to the region of the gene as well asthose of the readswhich match in more places (below the limit set in thedialog in figure 18.110) that have been allocated tothis gene's region. Agene's region is that comprised of the flanking regions(if it was specified infigure 18.110), the exons, the introns andacross exon-exonboundaries of all trans annotated for the gene. Thus,the sum of the totalgene reads numbers is the number of mapped reads for thesample (you can findthe number in the RNA-Seq report).map的reads總和。映射到某個(gè)基因上的所有reads總數(shù)。因此這包含所有的映射到這個(gè)區(qū)域上的reads。
舉例:
比如對(duì)應(yīng)到該基因的read有1000個(gè),總reads個(gè)數(shù)有100萬(wàn),而該基因的外顯子總長(zhǎng)為5kb,那么它的RPKM為:10^9*1000(reads個(gè)數(shù))/10^6(總reads個(gè)數(shù))*5000(外顯子長(zhǎng)度)=200或者:1000(reads個(gè)數(shù))/1(百萬(wàn))*5(K)=200這個(gè)值反映基因的表達(dá)水平。
FPKM(fragmentsper kilobase of exon per million fragments mapped). FPKM與RPKM計(jì)算方法基本一致。不同點(diǎn)就是FPKM計(jì)算的是fragments,而RPKM計(jì)算的是reads。Fragment比read的含義更廣,因此FPKM包含的意義也更廣,可以是pair-end的一個(gè)fragment,也可以是一個(gè)read。
28、什么是轉(zhuǎn)錄本重構(gòu)
用測(cè)序的數(shù)據(jù)組裝成轉(zhuǎn)錄本。有兩種組裝方式:1,de-novo構(gòu)建; 2,有參考基因組重構(gòu)。其中de-novo組裝是指在不依賴參考基因組的情況下,將有overlap的reads連接成一個(gè)更長(zhǎng)的序列,經(jīng)過(guò)不斷的延伸,拼成一個(gè)個(gè)的contig及scaffold。常用工具包括velvet,trans-ABYSS,Trinity等。有參考基因組重構(gòu),是指先將read貼回到基因組上,然后在基因組通過(guò)reads覆蓋度,junction位點(diǎn)的信息等得到轉(zhuǎn)錄本,常用工具包括ure、cufflinks。
29、什么是表達(dá)譜
基因表達(dá)譜(geneexpression profile):指通過(guò)構(gòu)建處于某一特定狀態(tài)下的細(xì)胞或組織的非偏性cDNA文庫(kù),大規(guī)模cDNA測(cè)序,收集cDNA序列片段、定性、定量分析其mRNA群體組成,從而描繪該特定細(xì)胞或組織在特定狀態(tài)下的基因表達(dá)種類和豐度信息,這樣編制成的數(shù)據(jù)表就稱為基因表達(dá)譜。
30、什么是比較基因組學(xué)
比較基因組學(xué)(ComparativeGenomics)是基于基因組圖譜和測(cè)序基礎(chǔ)上,對(duì)已知的基因和基因組結(jié)構(gòu)進(jìn)行比較,來(lái)了解基因的功能、表達(dá)機(jī)理和物種進(jìn)化的學(xué)科。利用模式生物基因組與人類基因組之間編碼順序上和結(jié)構(gòu)上的同源性,克隆人類疾病基因,揭示基因功能和疾病分子機(jī)制,闡明物種進(jìn)化關(guān)系,及基因組的內(nèi)在結(jié)構(gòu)。
31、什么是基因組注釋
基因組注釋(Genomeannotation) 是利用生物信息學(xué)方法和工具,對(duì)基因組所有基因的生物學(xué)功能進(jìn)行高通量注釋,是當(dāng)前功能基因組學(xué)研究的一個(gè)熱點(diǎn)。基因組注釋的研究?jī)?nèi)容包括基因識(shí)別和基因功能注釋兩個(gè)方面。基因識(shí)別的核心是確定全基因組序列中所有基因的確切位置。